Une approche fondamentalement nouvelle pour explorer et classer la diversité fonctionnelle des séquences génomiques
En génomique, les séquences naturelles issues de milliards d’années d’évolution représentent un immense réservoir de diversité. Cependant, il reste encore peu exploré en raison, d’une part, des informations biochimiques, fonctionnelles et structurelles trop limitées dont on dispose et, d’autre part, d’un manque d’algorithmes puissants pour classifier les fonctions protéiques. Une équipe de recherche du Laboratoire de biologie computationnelle et quantitative (Sorbonne Université, CNRS), en collaboration avec le laboratoire Biologie du chloroplaste et perception de la lumière chez les microalgues (Sorbonne Université, CNRS), a développé une approche computationnelle inédite pour la classification fonctionnelle de familles protéiques. Présentée dans un article publié le 30 mars 2022 dans Molecular Biology and Evolution, cette approche vise à permettre la découverte de protéines jouant des rôles fonctionnels clés dans des domaines très variés tels que la biologie, la santé humaine ou les biotechnologies.
-
Alessandra Carbone, professeure d’informatique, directrice du Laboratoire du biologique computationnelle et quantitative (Sorbonne Université, CNRS)
-
Marion Valzy, service de presse de Sorbonne Université
-
06 14 02 20 51
-
Claire de Thoisy-Méchin, service de presse de Sorbonne Université
-
06 74 03 40 19
Le défi est de classer des milliers de séquences de protéines provenant du même ancêtre, possiblement très divergentes, qui sont impliquées dans l'interaction avec les acides nucléiques, les acides aminés et les petites molécules, et dont les fonctions doivent être découvertes ou caractérisées plus précisément. L’enjeu pour les chercheuses et chercheurs est de « lire » les différentes caractéristiques fonctionnelles directement à partir de leur séquence dans les génomes.
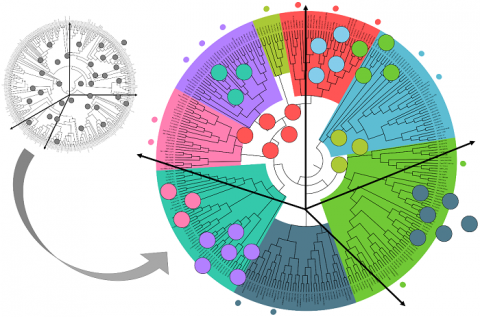
ProfileView (ou « Point de vue des profils ») est une approche computationnelle innovante, développée au Laboratoire de biologie computationnelle et quantitative (Sorbonne Université, CNRS) qui permet d'apprendre les différences fonctionnelles entre protéines et de découvrir de nouvelles séquences naturelles ayant des fonctions potentiellement intéressantes en biotechnologie. ProfileView est conçue pour classer des milliers de séquences ayant un ancêtre commun par fonction. Cette approche, et le logiciel qui en découle, s'appuient sur deux idées principales:
1. l'utilisation de multiples modèles probabilistes, appelés « profils », pour explorer et extraire de façon novatrice des informations évolutives de l'immense espace des séquences
2. une nouvelle définition d'espace de représentation des séquences où l'on regarde les séquences du point de vue des motifs fonctionnels encodés dans les profils.
ProfileView a été validée sur plusieurs familles de protéines, connues pour être difficilement annotées fonctionnellement. Il s'agit de classes de protéines très répandues dans l’environnement, présentant une grande variété de fonctions, et une forte divergence de séquences. Une de ces familles protéiques est celle des Cryptochrome-Photolyases dont les membres sont impliqués dans divers mécanismes biologiques activés par la lumière et étudiée au laboratoire Biologie du chloroplaste et perception de la lumière chez les microalgues (Sorbonne Université, CNRS). Des membres de cette famille de protéines sont extrêmement importants en médecine et biologie en raison de leur rôle clé dans la stabilité du génome, la biologie du cancer, la régulation des rythmes circadiens ou encore les méthodologies optogénétiques. Les résultats de ProfileView appuient un vaste ensemble de connaissances correspondant aux travaux expérimentaux existants et résolvent des classifications fonctionnelles encore non définies. Ce retour d'expérience montre que ProfileView permet une approche puissante pour collecter des informations sur la diversité fonctionnelle des protéines, de sélectionner des séquences en vue de la conception d'expériences fonctionnelles ciblées, et de découvrir de nouvelles fonctions biologiques.
ProfileView offre un regard computationnel totalement nouveau sur les processus évolutifs et sur l'espace complexe des séquences naturelles. Cette approche permet une classification très générale et précise des membres des familles de protéines, mettant en évidence les motifs fonctionnels d'interaction avec d'autres protéines, l'ADN et les petites molécules, et ouvrant la voie à des analyses à grande échelle. Ses propriétés visent ainsi à permettre la découverte de protéines jouant des rôles fonctionnels clés dans des domaines très variés tels que la biologie, la santé humaine ou les biotechnologies.
Pour en savoir plus :
ProfileView est un logiciel libre. Le logiciel et les données générées dans cette étude sont disponibles en ligne.
Référence :
Vicedomini, R., Bouly, J. P., Laine, E., Falciatore, A., & Carbone, A. (2022) Multiple probabilistic models extract features from protein sequence data and resolve functional diversity of very different protein families. Molecular Biology and Evolution, msac070, https://doi.org/10.1093/molbev/msac070
